Por: Fred Furtado
Enviar a un amigo
Los detalles proporcionados en esta página no serán usados para enviar correo electrónico no solicitado y no se venderán a terceros. Ver política de privacidad.
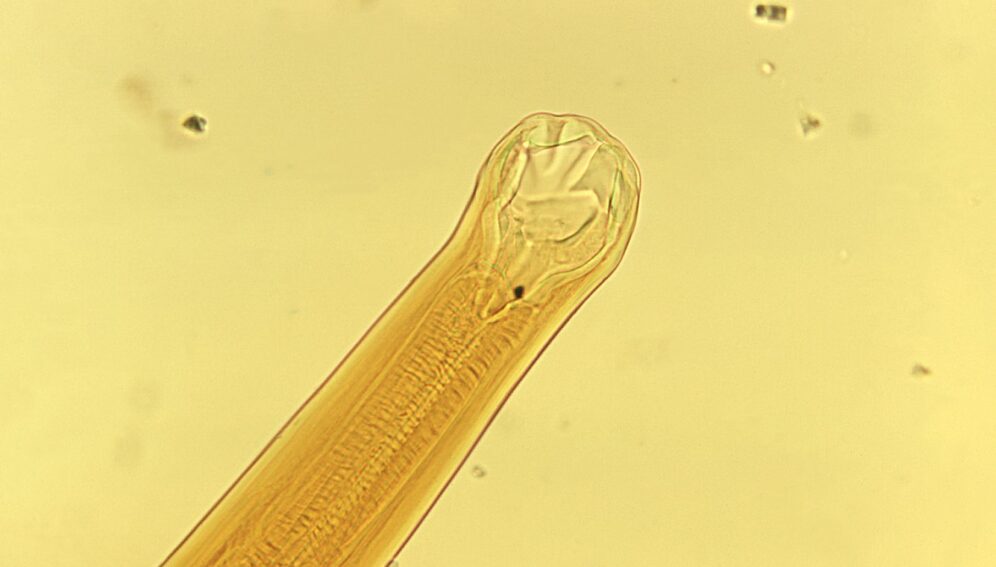
Científicos lograron secuenciar el genoma del Necator americanus —parásito responsable de aproximadamente el 85 por ciento de las infecciones de anquilostoma en humanos— con lo cual hoy tienen una visión sin precedentes de la biología del gusano, lo que podría ayudar a acelerar el desarrollo de medicamentos, diagnósticos y vacunas para combatirlo.
Los científicos también usaron la secuencia del genoma del anquilostoma para identificar posibles objetivos para medicamentos y vacunas, y publicaron sus hallazgos en Nature Genetics esta semana (19 de enero).
Los anquilostomas son responsables de una de las enfermedades tropicales olvidadas que afecta a 700 millones de personas de comunidades pobres. La infección con N. americanus conduce a la anemia, desnutrición en mujeres embarazadas y a deficiencias en el desarrollo cognitivo y físico de los niños.
Debido a que el fracaso del tratamiento por la resistencia a los fármacos se ha convertido en un reto para las terapias actuales contra la enfermedad, se requieren nuevas intervenciones, señala el artículo.
Pero la carencia de la secuencia genética completa del parásito obstaculizaba la búsqueda de nuevos enfoques, dicen los científicos.
Un ejemplo es una familia de proteínas conocida como SCP/TAP, que participa en las interacciones huésped-parásito.
“Fueron estudiadas como candidatas potenciales para el desarrollo de tratamientos”, revela la autora, Makedonka Mitreva, del Instituto Genoma de la Universidad de Washington, Estados Unidos.
“Sin embargo, el complemento total y la complejidad de su familia de genes no se conocía”, añade.
El estudio identificó 96 de esas proteínas específicas de N. americanus, que podrían ser objetivos potenciales de medicamentos o vacunas.
Stefan Geiger, inmunólogo de la Universidad Federal de Minas Gerais, Brasil, se muestra complacido por el estudio.
“Esta información revelará las funciones esenciales del parásito y permitirá el diseño de sustancias muy necesarias para los seres humanos que [actúan específicamente] contra este organismo”, asegura.
Los investigadores también usaron los nuevos datos sobre las proteínas quinasas del anquilostoma, un grupo de enzimas que regula muchos procesos celulares, para examinar los fármacos existentes y los inhibidores de los fármacos potenciales. La sustancia de mayor puntuación fue una molécula aprobada para el tratamiento de una forma de leucemia, pero otros 233 compuestos también probaron ser prometedores, según el artículo.
La identificación de medicamentos ya existentes que también se pueden usar para tratar el anquilostoma es una forma barata de obtener sustancias que se conviertan en fármacos muy necesarios, para ser usados especialmente en los países desarrollados, subraya Mitreva.
“La decodificación del genoma del Necator americanus es solo el comienzo de la era posgenómica en la investigación del anquilostoma”, añade.
Los investigadores también ofrecieron un ejemplo de cómo la secuenciación del genoma se puede usar ahora para desarrollar un mayor conocimiento del patógeno.
Crearon micro matrices de 564 proteínas de los códigos del genoma y los probaron con la sangre de 200 individuos del estado de Minas Gerais en Brasil. Esto les permitió identificar 22 antígenos —partes de esas proteínas— a los que respondieron los sistemas inmunológicos, así como mirar cómo los diferentes sistemas inmunológicos de las personas responden de manera diferente.
“Este enfoque es importante porque nos permite comparar las respuestas inmunológicas basadas en distintos perfiles de infección —como la edad, duración de la infección, carga parasitaria, historia de exposición y antecedentes genéticos— e identificar los diferentes antígenos”, explica Geiger.
Enlace al artículo en Nature Genomics
La versión original de este artículo se publicó en la edición global de SciDev.Net
References
Nature Genetics doi:10.1038/ng.2875 (2013)