Por: Paula Leighton
Enviar a un amigo
Los detalles proporcionados en esta página no serán usados para enviar correo electrónico no solicitado y no se venderán a terceros. Ver política de privacidad.
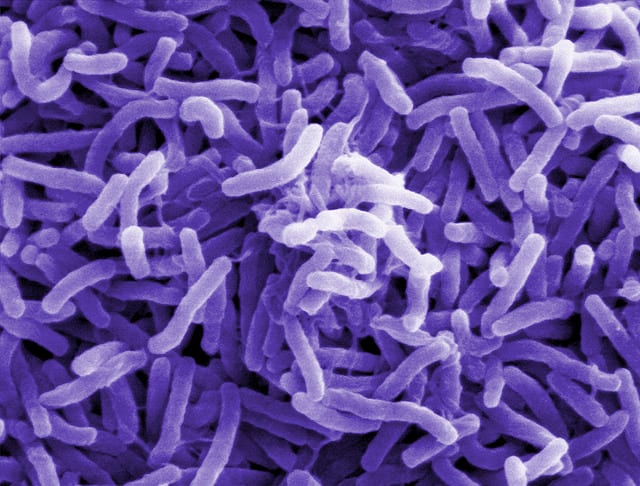
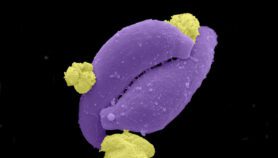
[SANTIAGO] Determinar el linaje del V. cholerae causante de un brote de cólera en América Latina permitiría diseñar estrategias más eficientes de control de la enfermedad, considerando como preocupaciones de salud pública solo aquellos casos que no corresponden a las cepas locales.
Así lo plantea un equipo internacional de investigadores en Science, luego de caracterizar 252 aislamientos de V. Cholerae causantes de brotes ocurridos entre 1974 y 2014 en 14 países latinoamericanos.
“No es bueno bajar la guardia. Mientras no haya 100 por ciento de cobertura de agua segura y ojalá tratamiento de aguas servidas, el riesgo de cólera seguirá siendo alto”.
Jorge Jiménez, Universidad Católica de Chile
Empleando la técnica de secuenciación de genoma completo, lograron determinar que solo las dos epidemias ocurridas en Latinoamérica —la originada en 1991 en Perú y la de 2010 en Haití— se debieron a cepas originadas en el biotipo pandémico El Tor (7PET) del V. cholerae, causante de la llamada “séptima pandemia”.
Este surgió en 1961 en Indonesia, desde donde se propagó a otros países de Asia, Europa y África, hasta llegar en 1991 a América Latina, que había estado libre de epidemias de cólera por más de un siglo.
“Estos linajes pandémicos son responsables de epidemias masivas y explosivas en un corto lapso” describe el estudio. La de Perú, por ejemplo, infectó a 1,2 millón de personas y dejó 12 mil fallecidos hacia 1997. En Haití, la epidemia infectó a más de 797 mil personas y causó más de 9.400 muertes.
En tanto, el análisis genómico mostró que por lo menos siete linajes de V. cholerae locales o autóctonos identificados en las Américas se asocian a dos patrones de enfermedad que son distintos al epidémico.
Un patrón se asocia a casos esporádicos o brotes limitados, donde prácticamente no hay infecciones secundarias, y el otro genera casos a lo largo de períodos prolongados y áreas geográficas más amplias (ej. 65 infecciones durante 20 años en la Costa del Golfo de EE.UU.).
Esto significa que hoy es posible reconocer las diferencias entre los linajes pandémicos y los locales “y seguirle la pista a la cepa más relevante para el cólera epidémico. Usar esta información permite priorizar esfuerzos de intervención a nivel de salud pública, lo que es especialmente importante cuando los recursos son limitados y los fondos deben maximizarse”, dice a SciDev.Net Daryl Domman, investigador del Wellcome Trust Sanger Institute (Reino Unido) y autor principal del estudio.

Y lo ilustra con un ejemplo: “En 2009 ocurrió un pequeño brote en Paraguay. Según los tests estándar de laboratorio, parecía cólera epidémico. Se elaboró una gran respuesta de salud pública en muchos países de la región temiendo que viniera un brote mayor. Por nuestro estudio ahora sabemos que ese brote no se originó en una cepa pandémica. Si lo hubiéramos sabido antes, la respuesta habría sido más mesurada”.
Jorge Jiménez, investigador del Instituto Milenio de Inmunología e Inmunoterapia, en Chile, quien en 1991 encabezó como ministro de Salud la campaña de Control del Cólera en este país, señala que transcurridos 26 años “el aporte de la biología molecular ciertamente aclara muchas cosas y la diferenciación entre cepas pandémicas y locales es consistente, cosa que de alguna manera se sabía, pero no se había podido demostrar”.
Domman enfatiza que “la secuenciación de genoma completo tiene la mayor resolución posible y entrega la mayor información” y, a medida que su precio siga bajando, “se volverá el estándar para tomar decisiones de salud pública ante la mayoría de las enfermedades infecciosas”.
Jiménez reconoce que identificar cepas más agresivas o menos agresivas generará cambios en el diseño de respuestas de salud pública, pero advierte que “no es bueno bajar la guardia. Mientras no haya 100 por ciento de cobertura de agua segura y ojalá tratamiento de aguas servidas, el riesgo de cólera seguirá siendo alto”.