Par: Claude Foly Akoussan
Envoyer à un ami
Les coordonnées que vous indiquez sur cette page ne seront pas utilisées pour vous envoyer des emails non- sollicités et ne seront pas vendues à un tiers. Voir politique de confidentialité.
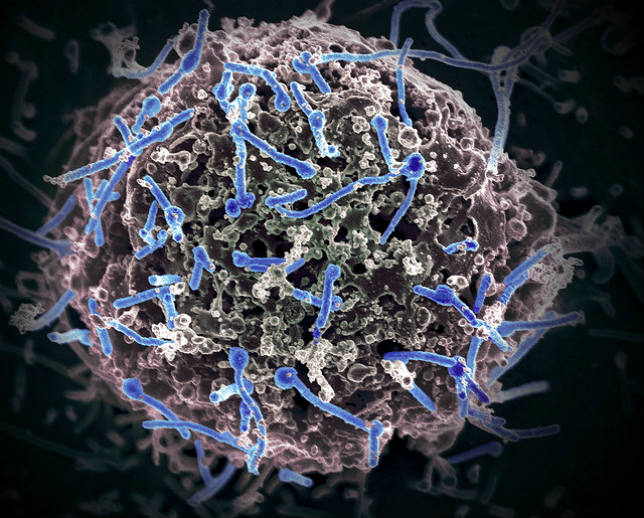
Des recherches menées entre autres par l'Institut Pasteur (de Dakar et de Paris) et l'Université de Sydney révèlent qu’il existe en Guinée trois variantes distinctes du virus Ebola.
La première variante dénommée "GUI-1" concerne les virus prélevés au début de l'épidémie en mars 2014 ; elle est présente seulement en Guinée, à la fois dans les zones urbaines et dans les régions forestières.
La deuxième variante (GUI-2) se rapporte aux virus identifiés en Sierra Leone ; tandis que la troisième variante (SLE-GUI-3) a été identifiée à Conakry et dans les villes proches comme Forécariah, Dalaba et Coyah.
La combinaison des données épidémiologiques et des similitudes notées entre cette variante et les virus de Sierra Leone montre qu’il y a eu plusieurs réintroductions du virus Ebola en provenance de Sierra Leone dans la région autour de Conakry.
Contacté par SciDev.Net, Etienne Simon-Lorière, membre de l’équipe de recherche de l’Institut Pasteur de Paris explique que "les recherches ont eu lieu essentiellement en Sierra Leone et en Guinée pour séquencer le virus en vue de retracer son cheminement et sa diffusion en Guinée, depuis la zone forestière qui est l'épicentre identifié vers la capitale Conakry."
"Le séquençage a été fait à partir d'échantillons rares contenant seulement de petites quantités de matériel biologique puis optimisé avec des données fournies par des chercheurs du Broad Institute de l’Université de Cambridge aux Etats Unis, qui étaient déployés en Sierra Leone", explique-t-il.
"Et nous nous sommes rendu compte que le virus présent dans la capitale guinéenne est venu aussi en partie de la Sierra Léone voisine. Ce n'est donc pas un seul type de virus qu'il y avait dans la capitale mais un mélange de trois virus", conclut-il.
“Ces résultats, mis à la disposition de la communauté scientifique, permettront d'optimiser les traitements et vaccins en cours de développement, surtout que c'est la première fois que le virus reste ainsi chez l'homme pendant une longue période.”
Etienne Simon-Lorière, chercheur à l'Institut Pasteur, Paris
A en croire les chercheurs, chacune de ces variantes se caractérise par une combinaison de mutations affectant différentes protéines virales : la protéine VP35 qui peut être un facteur de virulence ; la glycoprotéine de l'enveloppe, ce qui peut modifier la perception du virus par le système immunitaire ; la polymérase, qui est habituellement la région virale la mieux conservée.
Les chercheurs rappellent que la caractérisation des variations génétiques du virus est essentielle pour garantir l'efficacité continue des outils de diagnostic et pour le développement de traitements ou de vaccins efficaces.
"Heureusement, indiquent les chercheurs, les mutations constatées à Conakry ne se sont pas traduites, du moins pour le moment, par plus de virulence ou plus d’aptitude du virus à se transmettre."
Etienne Simon-Lorière ajoute même que "ces résultats, mis à la disposition de la communauté scientifique, permettront d'optimiser les traitements et vaccins en cours de développement, surtout que c'est la première fois que le virus reste ainsi chez l'homme pendant une longue période".
Depuis l’année dernière, l'Institut Pasteur de Dakar a mis en place un laboratoire mobile à l'hôpital Donka de Conakry, pour fournir des services de diagnostic.
"Il y a quelques mois, le séquençage prenait jusqu’à deux jours. Mais avec le déploiement sur le terrain de dispositifs de tests indépendants et mobiles qui n'ont pas besoin d'être dans un laboratoire très organisé, un échantillon peut être maintenant séquencé en une dizaine d'heures", précise Simon-Lorière.
Avec la persistance de l'épidémie et des cas qui émergent dans des endroits inattendus, ces séquençages vont peut-être contribuer à enfin atteindre l’objectif de zéro cas en Guinée et en Sierra Léone.
Depuis son apparition en mars 2014, l’épidémie de la fièvre Ebola qui a sévi en Guinée, en Sierra Leone et au Libéria a déjà pris fin dans ce dernier pays.
Selon les dernières statistiques de Médecins sans Frontières, elle a contaminé à ce jour plus de 25 000 personnes et fait plus de 10 000 morts.